Os eventos citológicos e as pressões seletivas na origem da meiose ainda estão sendo reconhecidos. Atualmente a proposta da biologia é que a meiose evoluiu a partir da mitose produzindo quatro novas etapas: o emparelhamento de cromossomos homólogos, a ocorrência de extensa recombinação entre as cromátides não-irmãs durante este emparelhamento, a supressão da separação da cromátide-irmã durante a primeira divisão meiótica e a ausência de replicação cromossômica durante a segunda divisão meiótica. A biologia molecular esta começando a entender como isto evoluiu.

Meiose (Telófase II) – Ao atingir os polos, os cromossomos descondensam-se e forma-se novamente um núcleo em torno de cada conjunto, formando quatro células haploides.
A origem simultânea de todos esses recursos em uma só etapa é improvável, igualmente é valido para uma origem conquistada passo-a-passo através da seleção de mutações, dado que a seqüência de eventos necessária para a produção de conjuntos cromossômicos haploides. Tanto Maynard Smith (1978) quanto Hamilton (1999) consideraram a origem da meiose como um dos problemas mais difíceis da evolução solucionar.
Isto não deveria assustar as pessoas. A origem dos eucariotos também foi (e ainda é, de certa forma) pregada como algo improvável devido aos níveis de organização da célula, mas na última década muitas evidências moleculares foram descritas e muitos grupos biológicos, reinos, supergrupos foram descobertos e descritos demonstrando que há evidências claras para um contexto evolutivo. A mesma coisa é com a mitose e com a meiose. Devemos lembrar também que a meiose só foi aceita como a teoria do cruzamento cromossômico a partir de Thomas Hunt Morgan, com base na descoberta de Frans Alfons Janssens que descreveu o fenômeno em 1909, designando-o por “quiasmatopia”.
O termo quiasma está intimamente ligado ao cruzamento cromossômico e Morgan percebeu a importância da interpretação citológica do quiasma de Janssens para a sua própria pesquisa sobre a hereditariedade da Drosophila sp. Os fundamentos físicos do cruzamento cromossômico foram demonstrados pela primeira vez por Harriet Creighton e Barbara McClintock somente em 1931. Desta forma, a meiose é compreendida pela biologia somente a poucos anos e sua origem e evolução ainda é pouco compreendida.
Uma das melhores referências científicas que há para caracterizar a origem e evolução (ou as origens) da meiose a partir da mitose é o trabalho de Adam S. Wilkins e Robin Holliday (2009) na qual traz uma perspectiva evolucionária relativamente completa.
As origens da meiose englobam tanto as novidades citológicas quanto forças seletivas que podem ter favorecido o evento.
Notavelmente, as primeiras espécies eucarióticas eram haploides e de célula única, possuindo apenas um único conjunto de cromossomos que se propagava via mitose. Atualmente os grupos mais simples de eucariotos são formados por protistas ou fungos que exibem uma propagação mitótica dos estados haploides (possui um único conjunto completo de cromossomos) e diploides (possui dois conjuntos completos de cromossomos). A diploidia é certamente um estado derivado, e a princípio, a primeira célula diploide poderia ter surgido por fusão celular ou por endomitose.
Endomitose é um processo de duplicação cromossômica dentro da membrana nuclear. Não ocorre a telófase nem citocinese (fases finais de uma divisão celular), tendo como resultado uma única célula, com o dobro dos cromossomos.
Esses primeiros diploides provavelmente surgiram através de erros endomitótico em vez de fusão celular. No entanto, a fusão de células não-sexuais e núcleos podem ocorrer independentemente do sexo. É o que a biologia chama de parassexualidade. Um exemplo disto ocorre com certos fungos onde hifas diferentes podem fundir seus núcleos que ocuparão a mesma célula em um evento chamado de plasmogonia. Sob este ponto de vista, a formação de células diploides ocasionais antecedeu ciclos de vida sexuais regulares em eucariotas.
O fato da mitose ser uma propriedade eucariótica universal sugere que este tipo de divisão celular surgiu na base da árvore eucariótica. Uma evidência muito importante é que todas as moléculas-chave empregadas na mitose eucariótica são homólogas aos procarióticos (Hirano 2005 e Erickson 2007). Isso inclui as actinas, necessárias para a separação de células filha em eucariotas; as tubulinas, que em eucariotos formam o fuso mitótico e o movimento dos cromossomos; e as moléculas necessárias para a condensação de cromossomos e coesão das cromátides-irmãs; membros da proteína structural maintenance of chromosomes (proteína de manutenção estrutural dos cromossomos, cuja sigla é SMC) uma família proteica. Nos membros procariotas que tem a família das tubulina há genes FtsZ, que foram descobertos pela primeira vez em Escherichia coli e que posteriormente foram encontrados em muitos outros procariotas, e seus respectivos homólogos SMC foram encontrados em todos os reinos eubacterianos e archaebacteriais.
Os genes da família FtsZ e SMC-actina são relacionados evolutivamente e podem ter sido recrutados para uso nas primeiras formas primitivas de mitose; envolvidos em pontos de fixação para segregar cromossomos irmãos.
Embora as várias semelhanças entre as duas formas de divisão celular demonstrem uma estreita relação evolutiva entre elas, a maior complexidade da meiose indica que ela é derivada do processo. Além disso, enquanto a mitose é universal em espécies eucariótas, a meiose é apenas presente, uma vez que algumas linhagens eucarióticas perderam sua capacidade. Evidências comparativas sugerem que a meiose apareceu no início da história célular eucariótica (Ramesh et al, 2005;. Schurko e Logsdon 2008), e seu alto grau de semelhança em diferentes grupos taxonômicos sugere que ela surgiu apenas uma vez (Hamilton 1999; Ramesh et al, 2005) .
Das quatro novas etapas da meiose citadas no início do texto, os geneticistas geralmente se focam na segunda (a ocorrência de recombinação eficiente intergênica entre homólogos durante emparelhamento), no entanto, enquanto a recombinação genética é uma característica fundamental da meiose, não é exclusiva para este processo. A capacidade de recombinação é encontrada em todos os procariontes e, portanto, deve preceder consideravelmente os eucariotos, e claro, a meiose (Levin, 1988).
A hipótese de que a recombinação meiótica surgiu da bacterial transformation (conversão bacteriana), partindo do princípio de que devido sua importância adaptativa, a evolução do sexo era um processo contínuo a partir de bactérias para eucariotas. Em biologia molecular, a conversão bacteriana é a alteração genética de uma célula que resulta da absorção e incorporação direta de material genético exógeno. A conversão bacteriana ocorre naturalmente em algumas espécies, mas também pode ser feita por meios artificiais em outras células. Para a conversão acontecer, as bactérias devem estar em um estado de competência, o que pode ocorrer como uma resposta por tempo limitado a condições ambientais, tais como a “fome” e a densidade celular. A conversão bacteriana, é semelhante a meiose, ocorre dentro de uma célula bacteriana, meio no qual cromossomos homólogos-não irmão (ou partes dele) se originam em células-mãe diferentes passando por recombinação genética. O DNA recombinado é passado para a progenie da bactéria. Os genes do núcleo para a meiose foram muito provavelmente presentes no ancestral comum de todos os eucariontes e a reprodução sexual envolvendo meiose é generalizada entre os eucariontes. A evidência deste processo sugere que eucariotos surgiram mais de 1,5 bilhões de anos atrás (Javaux et al, 2001). O evento crucial foi, provavelmente, o desenvolvimento de uma relação estável entre uma bactéria hospedeira anaeróbia e um menor, bactéria aeróbia internalizada. As cinco principais evidências que apoiam a hipótese de que a recombinação meiótica surgiu da conversão bacteriana são: 1) os genes fundamentais para a recombinação meiótica estavam presentes muito cedo na evolução eucariótica, possivelmente no ancestral comum de todos os eucariotas; 2) os ancestrais procarióticas diretos de eucariotas eram capazes de conversão; 3) ortólogos da recA, uma enzima que desempenha um papel central em conversão também desempenha um papel central na recombinação meiótica; 4) outras funções de genes (além dos ortólogos de recA) que são importantes para a meiose têm funções semelhantes na conversão bacteriana; e 5) a conversão bacteriana e de acoplamento de microrganismos eucarióticos (protistas) são induzidas por tipos semelhantes de stress que podem ser associados com danos ao DNA (Bernstein & Bernstein, 2010). Discutiremos mais sobre esta proposta mais adiante.
As células proto-eucarióticas originais devem ter possuído uma maquinaria enzimática para a recombinação. Em particular, um conjunto de moléculas cruciais para recombinação genética, que faz parte da família de proteínas recA, utilizada para a recombinação em ambos; procariotas e eucariotas (Aboussekhra et al, 1992). Veja mais aqui e aqui.
Em eucariotas, a recombinação genética não se restringe a meiose. Células somáticas diploides de fungos, plantas e animais são submetidos a um crossing-over cromossômico, um fenômeno conhecido como ”recombinações mitóticas”. Existem três contrastes significativos entre meiose e recombinação mitótica. Em primeiro lugar, a recombinação mitótica entre homólogos ocorre a uma frequência mais baixa do que a meiose. Em segundo lugar, enquanto o crossing-over entre cromátides-irmãs de células mitóticas é bastante frequente, na meiose está estruturado para promover o crossing-over entre cromátides não-irmãs. Em terceiro lugar, como encontrado em células de levedura, a recombinação mitótica é mediada eficazmente por qualquer um dos dois homólogos de recA; rad51 e Dmc1, enquanto que a troca meiótica entre homólogos requer especificamente Dmc1 (Neale e Keeney 2006). Assim, se a mitose precedeu a meiose em evolução, é igualmente provável que a recombinação mitótica precedeu a recombinação meiótica.
Um ponto interessante na origem da meiose é que ela não é um processo simplesmente concebido para a produção de células haplóides a partir de células diplóides. Em princípio, a replicação do DNA pré-meiótico não seria necessária. Os cromossomos não-replicados simplesmente emparelham um com o outro, com ou sem recombinação e migrariam para os polos opostos para produzir apenas dois núcleos haplóides. Todo o processo seria realizado em uma divisão, e não em duas.
Essa sequencia hipotética de eventos de um só passo da meiose (Cavalier-Smith 1981; Archetti 2004), difere dos ”dois passos” em que envolve: 1) a supressão ativa da síntese de DNA; e 2) o emparelhamento de cromossomos homólogos mas não replicados antes da metáfase.
A meiose atual começa com uma fase S, as quais podem diferir em certas características da fase S do ciclo mitótico (Stern & Hotta 1977), obtendo-se cromossomos constituídos por um par de cromátides-irmãs. Isto é seguido por emparelhamento de cromossomos homólogos ao longo de todo o seu comprimento (sinapse), um estado que é visivelmente evidente na maioria das espécies eucarióticas. Nesta fase, as cromátides irmãs não-homólogas recombinam-se uma com a outra, por vezes, apenas uma vez, mas mais frequentemente em vários locais ao longo do seu comprimento. Depois de recombinação, os cromossomos se condensam ainda mais e os homólogos emparelhados ficam alinhados na placa equatorial, esta é a metáfase.
Já existem dois cinetócoros ao qual estão ligados nas cromátides que, em contraste com a mitose, os cinetócoros não desmontados nesta primeira divisão meiótica: os homólogos simplesmente separaram-se para os polos opostos. Esta ausência de fissão do cinetócoro na primeira divisão meiótica (M1) reflete a diferença na mecânica molecular da fixação dos microtúbulos no centrômero em consequência da geometria da inserção da cromátide-irmã quando os homólogos estão emparelhados. Em contraste com a “bio-orientação” das cromátides irmãs para polos opostos na mitose, ambos as cromátides irmãs de cada cromossomo emparelhado na metafase I estão ligados com as fibras do fuso que funcionam ao mesmo como polo (mono-orientação) (Hauf e Watanabe 2004).
Quando os dois conjuntos de cromossomos produzidos na M1 são colocados dentro de núcleos, que já estão replicados. Então, esses núcleos ficam com um efeito equivalente ao estado G2 do ciclo mitótico. A ausência de replicação na segunda divisão meiótica (M2) presumivelmente segue a partir do mesmo mecanismo que impede novas rodadas de replicação na fase G2 das células em preparação para a mitose, isto é, a ausência de ligação de um ou dos vários ”fatores de licenciamento” (por exemplo, as proteínas Mcm 2-7) com as origens de replicação através da sua remoção durante a fase S (Blow & Dutta, 2005). Embora o mecanismo exato ainda não seja conhecido, parece provável que a separação da cromátide- irmã no centrômero gera um sinal que se inicia o processo de ”licenciamento replicação”. Na ausência dessa transição molecular, no final da meiose I, as cromátides não podem ser submetidas a uma nova volta de replicação.
Seja qual for o mecanismo que inibe uma segunda fase S, a prófase da meiose II consiste simplesmente na condensação do cromossomo. Isso, por sua vez, é seguido pela metáfase II e, em seguida, pela anáfase II, em que as cromátides-irmas são separadas e segregadas para polos opostos, dando origem a dois núcleos haploides somente com as (não-replicados) cromátides. A separação de cromátides-irmãs na M2 envolve atores e processos moleculares semelhantes aos envolvidos na separação das cromátides-irmãs na mitose (Rivera e Losada 2006). No seu conjunto, esta divisão produz uma tétrade de produtos a partir de cada núcleo meiótico inicial, e cada um destes núcleos filhos-finais possui um genoma (haploide) não replicado. A evolução da meiose em duas fases da meiose requer menos eventos novos do que os simples processos da primeira etapa. Necessita apenas da sinapse de cromossomos homólogos, cada um consistindo de duas cromátides-irmãs, o resto da sequência é o reconhecido padrão da mitose para cromossomos replicados.
A origem da meiose envolveu a evolução de uma sinapse estável no genoma, durando a metáfase, e a inserção deste passo no ciclo mitótico. Tal emparelhamento no início pode parecer uma novidade impressionante. No entanto, o emparelhamento dos homólogos nas células somáticas (não-meiótica) foi encontrado tanto em Drosophila (McKee, 2004) quanto em levedura (Burgess et al, 1999). Tal emparelhamento somático difere da sinapse meiótica em três aspectos: 1) não é tão extenso (McKee 2004); 2) não conduz a níveis de recombinação genética vistos na meiose; e 3) termina em qualquer interfase ou prófase, permitindo que cada cromossomo avance para a placa equatorial metafásica independentemente do seu homólogo.
A sinapse meiótica comporta-se como uma modificação de uma propriedade já existentes, não totalmente nova. A ausência de separação das cromátides-irmãs na M1 pode refletir uma geometria alterada de fixação dos microtúbulos, quando cinetócoros homólogos estão emparelhados, enquanto que a ausência de fase S na M2 seria uma consequência da ausência da separação das cromátides-irmã. A característica distintiva da meiose, ou seja, os níveis elevados de recombinação durante o emparelhamento cromossomo, podem então ser vistos como uma propriedade que evoluiu depois.
A ideia de que uma inovação-chave converteu um ciclo mitótico em um meiótico não é a primeira sugestão de seu tipo. Cavalier-Smith (2002) argumentou que a supressão da divisão do cinetócoro em M1 foi a principal inovação na meiose. Este evento, no entanto, vem após o emparelhamento homólogo, o que claramente é uma novidade. Além disso, como dito acima, a ausência de divisão de cinetócoro reflete diretamente a diferença na orientação da cromátide-irmã em relação aos polos entre M1 e M2 (Hauf e Watanabe, 2004). Este, por sua vez, reflete as diferenças estruturais (geométricas) inerentes aos microtúbulos e entre cromossomos pareados e não pareados ao nível dos cromossomos individuais (Paliulis e Nicklas, 2000).
A meiose originou-se com a inserção da sinapse em cromossomos homólogos no ciclo mitótico, e isto levanta muitas considerações. Uma delas diz respeito à natureza das pressões seletivas para este novo comportamento cromossômico, e outra, diz respeito aos requisitos moleculares para esta nova característica citológica.
De fato, existem benefícios seletivos provenientes da recombinação intergênica, são basicamente dois: ajuda a reduzir combinações genéticas desfavoráveis e promove novas combinações favoráveis. Correspondentemente, acredita-se que a evolução da meiose tem se focado nas pressões de seleção para promover a eliminação de combinações de genes prejudiciais (Fisher 1930; Muller 1932; Maynard Smith 1978). Deste ponto de vista, qualquer seleção para homologia sináptica envolveria seleção para a melhoria da eficiência de recombinação genética mediada por tal emparelhamento.
Mas mesmo a recombinação intergênica como a primeira força seletiva benéfica para a origem da meiose é algo enigmático. A explicação invoca implicitamente um elemento de seleção de grupo no que diz respeito aos benefícios futuros para a população. No entanto, a seleção natural não pode operar com previdência. Assim, quaisquer que sejam os benefícios iniciais do emparelhamento cromossômico em proto-eucariotas, eles teriam que ser mais imediatos do que a promoção de uma recombinação intergênica.
Uma alternativa é que o benefício inicial da meiose foi reforçar o reparo aos danos no DNA via recombinação (Bernstein 1977; Bernstein et al, 1988). A necessidade de uma reparação eficiente de DNA é um pré-requisito básico e antigo das células, continuamente presente em células procariotas, e serviu originalmente para proteger as células precoces da entrada de radiação solar UV e outros agentes que danificam o DNA, bem como a dessecação (saiba mais sobre o DNA como dissipador termodinâmico: 1, 2, 3 e 4). Além disso, a recombinação de sequências homólogas fornece um modo eficiente de reparo do DNA. Em E. coli, por exemplo, a inativação de qualquer uma das funções essenciais de recombinação (a enzima recA ou recBC) aumenta muito a letalidade após exposição a irradiação com UV, apesar da presença de outros sistemas de reparação de DNA (Clark, 1971 & Smith, 2004).
O argumento para o reparo do DNA como o principal benefício da meiose implica que as formas existentes de reparo de DNA foram uma fronteira insuficiente para as necessidades das primeiras células eucarióticas. Procariontes, são dotados de uma rica variedade de capacidades de reparo do DNA, incluindo a reparação recombinacional induzível (Levin 1988).
A abundante vida procariótica nas duras condições de mares Arqueano (Knoll, 2003), bem antes da existência células eucarióticas, sugere que as capacidades de reparo de DNA devem ter bastado para lidar com os tipos de danos ao DNA associados a esse ambiente. Uma reparação eficiente de quebras de cadeia dupla (ou double-strand breaks, sigla em inglês DSBs) utilizando cromátides-irmãs em células mitóticas (Argueso et al, 2008), na meiose parece ter sido necessária para reparação do DNA onde uma propriedade adicional não parece ter sido necessária.
Se as sinapses entre homólogos foi o evento inicial chave na origem da meiose, isto teria gerado rendimentos a célula. Um alinhamento preciso entre os cromossomos pode ser a chave para o enigma, pois ele deve promover não só a recombinação, mas também a recombinação entre sequências longas inteiramente cobertas. É possível e bem provável que a sinapses entre os cromossomos homólogos foi selecionada porque promove a fidelidade de recombinação, reduzindo assim as chances de um emparelhamento deslocado e consequente recombinação. O tamanho do emparelhamento homólogo ajudaria a garantir que apenas as regiões idênticas (que não divergiram dos homólogos em diferentes localizações cromossômicas) se recombinem. Tal como na hipótese de reparação do DNA, os benefícios seletivos seriam imediatos, mas a vantagem proposta seria radicalmente diferente: em vez da restauração de sequências de DNA, o benefício seletivo deste novo processo seria a prevenção dos danos gerados por recombinação. Isto sugere que a recombinação, particularmente em células de multi-cromossômicas pode ter efeito deletério de tal modo que é regulado para minimiza-los (Bernstein et al, 1988). Há evidências experimentais diretas para esta proposição. Holliday et al, (1976) apresentaram uma extensa análise de mutantes defeituosos na reparação de DNA para um gene que eles designaram como rec-1 presente no fungo Ustilago maydis. O fenótipo destes mutantes, no entanto, é mais complexo do que uma simples deficiência de reparação. As estirpes apresentaram: 1) 20% de células não viáveis, 2) taxas elevadas de recombinação mitótica, 3) meioses defeituosas de cruzamentos entre linhagens diferentemente marcadas com produtos meióticos aneuploides e não viáveis, e 4) uma considerável heterogeneidade em células diploides, mas não em colônias haploides.
Este fenótipo complexo é mais visto como uma anormalidade na regulação (ou controle) de recombinação que se manifesta tanto na mitose quanto na meiose. A heterogeneidade dos diploides de das enzimas rec-1 são provavelmente devido a acontecimentos genéticos anormais gerados por recombinação e levando genomas ao desequilíbrio.
As enzimas recombinogênicas são normalmente mantidas a níveis baixos de atividade. A rad51 é homóloga da enzima recA e em Tetrahymena thermophila está normalmente presente a um baixo nível de atividade, mas após a exposição das células a UV ou metanossulfonato de metila (Campbell e Romero 1998), seus níveis aumentam dramaticamente, presumivelmente para facilitar a reparação recombinatória. Esta descoberta sugere que as atividades das enzimas de recA, em vez de emparelhamento homólogo, podem ser passos limitantes da velocidade para a recombinação. A recombinação tem de ser fortemente regulada, presumivelmente para evitar defeitos deletérios (Lynch, 2005).
Algumas aberrações cromossômicas produzidas por DSBs ocorrem preferencialmente em sequências repetitivas no genoma (Argueso et al, 2008). É provável que a crescente dimensão e complexidade genômica, uma característica fundamental da evolução eucariótica (Cavalier-Smith, 1978), tenha aumentado as oportunidades de eventos de recombinação entre tais sequências parálogas repetitivas em diferentes localizações cromossômicas. As consequências poderiam incluir deleções, duplicações, inversões na recombinação intra-cromossômica, intercâmbio intercromossômicos de cromossomos dicêntricos. O número de defeitos seria de esperar aumentar exponencialmente em função do aumento das sequências repetitivas ao longo do genoma. Tais alterações reduziriam a fidelidade da transmissão do genoma, e, consequentemente, a fração de células viáveis em qualquer linhagem. Em contraste, a recombinação na sinapse de homólogos deve reduzir substancialmente essa carga de dano induzido por recombinação. Ele não elimina totalmente, mas reduz substancialmente. Erros recombinacionais ocorrem na meiose, mesmo entre seqüências totalmente homólogas. Isto foi visto pela primeira vez em um fenômeno de deleção-duplicação de mutações (bar e supra) em Drosophila (Sturtevant, 1925). Como tais erros de recombinação são comuns, mesmo com o emparelhamento de homólogos na meiose o ponto-chave é que, na ausência de um emparelhamento preciso, tais erros ocorrem com ainda mais frequência. Por exemplo, a região macho-específico do cromossomo Y (MSY para a sigla em inglês de male-specific Y), que não tem parceiro de emparelhamento no X, parece ter acumulado um enorme estoque de duplicações permanentes e palíndromes como resultado de erros de recombinação entre as suas próprias sequências (Rozen et al, 2003; Skaletsky et al, 2003). Da mesma forma, sequências homologas imperfeitamente emparelhadas dentro de uma estirpe haploide derivada da espécie Brassica napus, que é uma espécie alopoliploide, sofrem muito mais recombinação com sequências não relacionadas da estirpe progenitora (Nicholas et al, 2007). A inferência razoável de todas essas descobertas é que na evolução da célula eucariótica inicial, qualquer tendência de aumento do tamanho do genoma através da adição de novas seqüências repetitivas teria aumentado a freqüência de erros de recombinação entre tais sequências.
Em células eucarióticas contemporâneas, tais eventos são evitados através do uso de checkpoints (pontos de checagem) no DNA onde há controle de danos, que implica em uma parada nas separações cromossômicas até que o reparo seja feito. Entretanto, células proto-eucariotas podem não ter tido estes checkpoints para fazer tais manutenções. Células procariotas contemporâneas parecem não ter estes mecanismos na conclusão da replicação (Bendich, 2007), e poderiam ter sido vulneráveis a tais erros de disjunção nos cromossomos.
Assim, as primeiras células diplóides proto-eucariotas tiveram que enfrentar este dilema. Elas teriam exigido reparação recombinacional eficiente para sua sobrevivência, mas teria tido necessidade de evitar os potenciais e concomitantes moldes de reparação, ou seja, erros recombinacionais entre sequencias não idênticas ou eventos de recombinação não resolvidos no momento da mitose.
Qualquer processo que promova o alinhamento preciso e recombinação de sequências de DNA restringe-se a um período diferente antes da separação dos cromossomos e ajudaria a resolver este dilema. Isto é precisamente o que o emparelhamento de homólogos na meiose consegue fazer. Ao realizar o emparelhamento, ocorre um exame cuidadoso reduzindo assim o número de adições, deleções e de sequencias de DNA aleatórias. Ao mesmo tempo, a concentração de eventos de recombinação para um período que precede a segregação cromossomo, como ocorre na sinapse dos homólogos, iria favorecer a manutenção da integridade genômica, através da redução de eventos de disjunções cromossômicas e, portanto, favorecendo a fidelidade da transmissão do genoma. Isso melhoraria a precisão recombinacional e a limitaria a um intervalo seguro, mantendo reparações benéficas. A linhagem celular que evoluiu essa capacidade para células diploides seria menos propensa a erros na transmissão de seu material genético. Otimizações subsequentes poderiam incluir atividades de enzimas que melhoraria a recombinação durante o período de emparelhamento do cromossomo e reduzir intervalos, como pode ser visto em células mitóticas normais.
Dentro deste molde, então, a sequência inicial proposta de eventos não contaria com a união de células sexuais ou processos parassexuais. Existe ainda um outro lado da moeda no que diz respeito ao cenário molecular da meiose.
A complexidade molecular e citológica do processo de emparelhamento esta presente nas espécies atuais (Kleckner, 2006) e a primeira aparição impossibilitou a origem de sinapses através de uma ou duas mutações na evolução das coesinas rec8 específicas de meiose e de um coesina preexistente (Parisi et al, 1999) foi sem dúvida um elemento crucial. Outras características citogenéticas, tais como os complexos sinaptonêmicos e a exigência de recombinação para promover a disjunção cromossômica normal poderiam muito bem ter evoluído posteriormente. Inicialmente, o emparelhamento simples em células diploides, contendo talvez apenas um ou dois pares homólogos, poderia ter envolvido menos componentes e passos. A evolução molecular de uma nova molécula como a coesina teria promovido emparelhamento de homólogos e fornecido o gatilho crucial para a meiose. Em células de levedura contemporâneas, a coesina rec8 é mantida especificamente em centrômeros e nas regiões adjacentes durante a sinapse normal dos homólogos que é essencial; sua ausência leva à perda da divisão, de redução e a ocorrência de separação das cromátides-irmãs (divisão equacional) em M1 (Watanabe & Nurse, 1999)
Alternativamente, é possível que a sinapse de homólogos foi inicialmente produzida por elevados índices de quebra cromossômica e junções, mediadas por sequencias homólogas teriam sido promovido pela coesinas existentes. Embora a sinapse de homólogos não exija DSBs em todos os organismos contemporâneos (Joyce & Kim, 2007) não poderia ter se envolvido nas primeiras formas de sinapse, em proto-eucariotas com um pequeno número de cromossomos, como a indução de recombinação pode, em princípio, ter bastado para iniciar o emparelhamento homólogo.
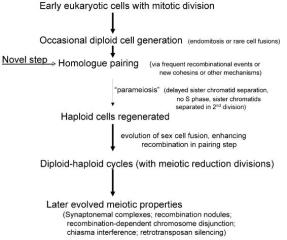
Esquema como uma linha de tempo de eventos na evolução da meiose. Setas grossas indicam eventos de longo prazo (escala de tempo evolutiva ou multi-geração), enquanto a seta fina representa processos de parameiose indicando uma consequência imediata. Clique para ampliar
As origens da sinapse de homólogos não pode ser conhecida com certeza, mas surpreendentemente a maquinaria molecular envolvida no processo é muito bem conservada entre procariotas e eucariotas e entre mitose e meiose. Em particular, o envolvimento de enzimas de recombinação, como vimos, a família da recA e seu enriquecimento em eucariotas atuais nos locais de recombinação durante meiose (Bishop, 1994; Tarsounas et al, 1999) que é uma evidência de continuidade evolucionária. A evolução molecular de Dmc1 foi claramente um passo fundamental na promoção da recombinação inter-homólogos, e como membro da família do gene recA, suas origens não são problemáticas.
Surpreendentemente, algumas das proteínas da família SMC, em particular as condensinas e as coesinas desempenham papéis semelhantes no controle do comportamento das cromátides-irmãs em ambas; meiose e mitose (Haering & Nasmyth, 2003). A maquinaria molecular para centrômero é compartilhada na mitose e M2. Estas moléculas incluem uma fosfatase de serina/treonina, PP2A e um dos seus substratos, a proteína-Shugoshin-associada-ao-cinetocoro (Rivera & Losado, 2006). Em resumo, é possível notar que a maioria dos componentes moleculares necessários para a evolução de emparelhamento entre homólogos e recombinação estão presentes em uma ou outra forma nas células eucarióticas proto-pré-meióticas.
A meiose esta intimamente ligada à reprodução sexual. De fato, ciclos de reprodução sexual seriam impossíveis sem a divisão celular reducional que ocorre na meiose. Para Wilkins e Holliday (2009) o advento evolutivo do emparelhamento de homólogo para eventos de diploidização podem ter ocorrido independentemente da fusão de células gaméticas. Tais eventos de diploidização, seguidos de recombinação e redução divisional para regenerar estados haploides, são denominados ”ciclos parassexuais”. Estes ciclos parassexuais foram descritos pela primeira vez em fungos (Pontecorvo, 1959) e permanecem como o mais caracterizado pela ciência, mas também são reconhecidos em células cancerosas tetraplóides, onde a redução de ploidia é de tetraploidia para diploidia (Rajamaran et al, 2005).
Wilkins e Holliday (2009) propõem que a sinapse entre homólogos e a concomitante redução dos estados diploides se originou em alguma forma de ciclo parassexual na linhagem proto-eucariota ancestral e que a relação funcional entre diploidização através da união de células sexuais e meiose foi um evento evolutivo subsequente. Sob ponto de vista, alguma forma de ”parameiose” (Becker & Castro-Prado, 2006) – a redução divisional de maior ploidia para um nível inferior sem uma fusão de células sexo anterior – precede a verdadeira meiose. A possibilidade de uma dissociação evolutiva entre eventos precoces de diploidização (e suas seqüelas redução/divisão) e meiose é consistente com o fato de que, do ponto de vista do desenvolvimento, a diploidização e meiose pode ser desacoplada. Em muitos eucariotas unicelulares, fusão de células haploides levam prontamente a fusão nuclear que imediatamente desencadeia a meiose, regenerando assim o estado haplóide. Em contraste, em eucariotos mais complexos e multicelulares, a meiose é grandemente retardada após a fusão inicial das células sexuais, ocorrendo muito mais tarde no ciclo de vida, durante a gametogênese. Obviamente, os diferentes sinais em diferentes organismos desencadeiam o aparecimento da meiose e em particular utilizado refletem a história evolutiva do organismo. Desta forma, alguma forma de divisão de redução poderia ter precedido ambas; a verdadeira meiose e os primeiros sistemas de união de células em sexo (unicelulares) eucariotas iniciais, como também sugerido por Hurst e Nurse (1991).
Conversão bacteriana e eucariogênese (recA, rad51 e Dmc1)
Protistas que são agentes patológicos em humanos divergiram muito cedo na linhagem eucarióta, e pressumivelmente eram assexuados. No entanto, vários destes organismos têm demonstrado a capacidade (ou ja tiveram a capacidade) de realizar a meiose e sexo. Por exemplo, Giardia intestinalis, um protozoário parasita intestinal comum, já foi considerado uma linhagem divergente precoce de eucariotas que representa um estágio de evolução eucariótica anterior ao surgimento da meiose. No entanto, a presença de um conjunto de cinco genes específicos de meiose, juntamente com outras evidências recentes, indicam que a G. intestinalis é capaz de fazer a meiose e, por conseguinte, a reprodução sexual (Ramesh et al, 2005, & Poxleitner et al, 2008). Protozoários parasitas do gênero Leishmania, que causam doenças humanas generalizadas, tem um ciclo sexual (Akopyants et al, 2009). Trichomonas vaginalis, um protista parasita que causa uma série de infecções em seres humanos e que acredita-se ter divergido da linhagem eucariótica bem no inicio da evolução deste grupo celular não faz meiose e nem sexo, no entanto, tem ortólogos de 27 dos 29 genes reconhecidos como funcionais na meiose; incluindo oito dos nove genes testados que são específicos para a meiose (Malik et al, 2008). Estes achados sugerem que o T. vaginalis é atualmente, ou se tornou recentemente sexuado. Esta observação e outras descobertas relacionadas levou à conclusão de que os genes necessários para a meiose do núcleo provavelmente estavam presentes no ancestral comum de todos os eucariotas (Malik et al, 2008).

Semelhanças da conversão bacteriana e recombinação meiótica eucariotas. DNA de cadeia simples (DNAss). Clique para ampliar
Estudos filogenéticos sugerem que 630 genes (ou mais) foram transferidos a partir do genoma de uma α-proteobacterial (a bacteria que deu origem as mitocôndrias) para o genoma nuclear eucariótica (Gabaldon e Huynen 2003). Portanto, se um ancestral α-proteobacterial era capaz de fazer algo semelhante a conversão bacteriana com este ancestral, assim como alguns dos seus parentes ainda existentes, seus genes que codificam as proteínas necessárias para a conversão podem ter sido integrado no genoma nuclear dos eucariotas.
Nos organismos ainda existentes, a família de genes de recA, rad51 e Dmc1 desempenham um papel central na recombinação meiótica (como vimos acima) e na conversão bacteriana. Os genes de recA detectados em eucariotas mostram um nível elevado de semelhança de sequências com α-proteobacteria, e tem sido sugerido que a transferência endossimbiótica do gene de recA tenha ocorrido a partir das primeiras mitocôndrias para o genoma nuclear de eucariotas ancestrais (Lin et al, 2006). Assim, é possível que um gene essencial para a recombinação meiótica tenha se originado de um gene bacteriano central para a conversão bacteriana.
O papel da recA na conversão bacteriana é essencial para a conversão em B. subtilis e em Streptococcus pneumoniae (Claverys et al, 2009). A expressão do gene de recA ocorre durante a recepção de DNA exógeno (também chamada de competência) em B. subtilis (Cheo et al, 1992) e S. pneumoniae (Mortier-Barriere et al, 1998). Neste processo, em B. subtilis, a entrada do DNA de cadeia simples (DNA single-stranded ou DNAss) e proteína recA interagem para formar estruturas filamentosas marcantes que emanam do polo celular contendo a maquinaria de competência e se estendendo para o citosol (Kidane e Graumann 2005). Estas estruturas representam nucleofilamentos dinâmicos de recA/DNAss que fazem a varredura do cromossomo residente em regiões de homologia trazendo inteiramente o DNAss para a região correspondente em que a troca ocorre (Kidane e Graumann 2005). Da mesma forma, a α-proteobacteria Helicobacter pylori é naturalmente competente para a conversão por um processo que emprega recA (Fischer e Haas 2004).
A reparação recombinatória não se limita à transformação de bactérias ou meiose em eucariotos. Também pode ocorrer com uma frequência inferior entre os cromossomos irmãos durante o crescimento vegetativo de bactérias e nas células somáticas de eucariotas.
Ainda sim, poderia-se argumentar que a função de recA em transformação e recombinação meiótica evoluíu de forma independente em células vegetativas de bactérias e eucariotas. No entanto, considera-se que a explicação mais simples é que recA que já estava ativamente envolvido no reparo durante o processo recombinacional de conversão e que a sua função no processo da meiose sexual era somente uma continuação do seu papel natural.
Assim, em eucariotos, o papel dos ortólogos da recA culminou na maquinaria de recombinação de cromossomos homólogos, um mecanismo de ação e regulação altamente conservado. Os genes rad51 e Dmc1 nas leveduras eucarióticas (Saccharomyces cerevisiae) e Schizosaccharomyces pombe são ortólogos de recA bacteriana. O gene Dmc1 também foi identificado em protistas tais como a Giardia, Trypanosoma, Leishmania, Entamoeba e Plasmodium (Ramesh et al, 2005). As rad51 e Dmc1 interagem com o DNAss para formar um intermediário filamentoso chamado de “filamento pré-sináptico”, que inicia a recombinação (Sauvageau et al, 2005, San Filippo et al, 2008). Considerando que a Dmc1 atua somente durante a meiose, rad51 atua tanto na meiose quanto reparo do DNA somático e durante a meiose utiliza principalmente a cromátide-irmã como o modelo de reparação; já Dmc1, usa predominantemente o cromossomo homólogo. Rad51 de levedura, assim como a recA bacteriana catalisa ATP (trifosfato de adenosina) no emparelhamento da fita de DNA com o homólogo (Sung, 1994). A Dmc1 tem uma similaridade global com a versão bacteriana recA, especialmente na estrutura terciária (Story et al, 1993). Estas observações sugerem que a rad51 e Dmc1 de leveduras (que atuam na meiose) e a recA bacteriana (que atua na conversão) são semelhantes em função e estão de acordo com a ideia de que a recombinação meiótica pode ter evoluído da conversão bacteriana. Sendo que os ortologos de recA tem a função na meiose em ratos, seres humanos, galinhas, nematóides, Arabidopsis, arroz e lírios. Em ratos, o ortólogo de recA é expresso a um nível bem elevado nos testículos e ovário, sugerindo que o produto proteico deste gene está envolvido em recombinação meiótica (Shinohara et ai, 1993).
Mutações no Dmc1 de ratos causam esterilidade, pois ocasiona a incapacidade de completar a meiose devido falhas de emparelhamento entre cromossomos homólogos (Pittman et al, 1998, Yoshida et al, 1998). Nos núcleos meióticas do nemátodo Caenorhabditis elegans, a resistência a danos no DNA causadoso por radiação X depende do gene rad51 (Takenami et al, 2000).
A expressão do gene ortólogo de recA também ocorre nos testículos e dos ovários de galináceos e nos testículos humanos. As formas humanas específicas da meiose do complexo Dmc1 sobre DNAss promove o intercâmbio de cadeias de DNA, onde as duas etapas essenciais da recombinação genética são homólogas (Sehorn et al, 2004, Bugreev et al, 2005).
No lírio, os genes lim15 e rad51 são ortólogos dos genes Dmc1 e rad51 de leveduras. Proteínas rad51 e lim15 de lírios se manifestam nos cromossomos em diferentes fases da prófase da M1 (Terasawa et al, 1995).
Em geral, recA e seus ortologos desempenham papéis muito semelhantes nos eventos de recombinação durante a transformação e meiose. Em ambos os casos, ela e seus equivalentes em DNAss formam um filamento pré-sináptico que capta uma molécula de DNA duplex e busca homologias estre si. Após a localização de homologia na molécula duplex, a molécula de pré-sináptica é capaz de formar articulações de DNA. Juntas elas, conduzem a recombinação genética que favorece tanto o reparo a erros quanto a recombinação genética.
Victor Rossetti
Palavras chave: NetNature, Rossetti, Meiose, Mitose, Recombinação genética, Cromossomos homólogos, Reparo de DNA, RecA, Rad51, Dmc1, Conversão bacteriana, Origem do sexo, Evolução.
.
Referências


É verdade essa informação que os criacionistas estão divulgando que o somos 50% bananas (http://www.criacionismo.com.br/2015/05/seres-humanos-sao-50-bananas.html)?
Bom, só se eles forem descendentes das bananas…embora acho que eles prefiram ser descendentes do pó!!!
Já escrevi sobre o assunto! https://netnature.wordpress.com/2012/08/22/uma-banana-para-os-criacionistas/
A afirmação de que somos no texto não tem artigos que suportam. Se observar as referências há textos sobre bananas. Um deles é um artigo da revista Nature, o outro não. Mas este texto (que não é um artigo) faz uso do mesmo artigo da Nature para suportar a ideia de que somos 50% bananas.
Se voce consultar o artigo original, ele não diz absolutamente nada sobre sermos 50% bananas. A ideia de que somos 50% bananas foi uma criação mal-intencionada dos criacionistas.
O artigo da Nature fala o exato oposto, mostra como houve duplicações no genoma da banana a 100 e depois a 60 milhoes de anos, e como ocorreu a domesticação da banana a 7 mil anos atrás.
Ué, como os criacionistas defensores da Terra Jovem ousam citar um artigo da Nature para falar que somos 50% bananas sendo que o artigo trata de duplicações genômicas e domesticação da banana ocorrido a mais de 6 mil anos e que, portanto, invalida a Terra jovem?? estranho não acha? rsrsrsr
Oi rosetti, quanto tempo.
Cara estava comentando um video e novamente um criacionista me aborda num “debate” sobre criacaçao e evoluçao, como ela já partiu apelando para os “evolucionistas” serem fanaticos e não aceitarem opinioes opostas; e depois ficou apelando para dizer que peer-reviiew não é valido, passando “fontes” (não)confiaveis como “darwinismo.wordpress”, entre outras.
Como minha area é biologia e não sou muito bom em geologia, nem em argumentaçao, gostaria de vers sobre esse assunto que os YEC estao falando agr:
http://gsabulletin.gsapubs.org/content/119/1-2/65.abstract
sério eles ficaram louquinhos por causa deste paper antigo e tão falando igual disco arranhado “ah a dataçao é falsa hurr durr” como sempre ficam pegando artigos cientificos fora de contexto e fazendo quote mining.
Mas como não sou um escritor tao bom quanto vc, nem sou muito bom em geologia gostaria de entender direito oque aconteceu.
Bem, deixa eu ver se entendi. Vou tentar explicar.
Este artigo estudou a geologia de três áreas separadas do Barberton Greenstone Belt (BGB) que mostram que o ironstone (um tipo de rocha sedimentar) foi depositado diretamente abaixo da superfície do solo moderno, em sistemas de águas subterrâneas durante os períodos de maior pluviosidade no Pleistoceno.
Ao que parece o estudo mostra que o ironstone depositado entre os blocos de sílex substituindo um tipo de quartzo e de outros 2 minerais; goetita e regolito. A predominância de goetita, que é um óxido de ferro termicamente instável esta bem presente na estratificação no local.
O problema é que essas áreas sofreram uma corrosão e representam uma relíquia do Arqueano quando ocorre esta substituição do oxido de ferro sob o quartzo. O ferro parece ter sido originado por unidades sedimentares sideríticas (mineral) do Arqueano e formaram altos topos em que ocorrem os corpos ironstone. A siderita foi dissolvida pela água subterrânea e o ferro depositado como óxidos de dentro do sistema de águas subterrâneas subsuperficial rasas onde a água rica em ferro fluiu para a superfície. Claro, este deposito, é um sistema hidrológico, mas como o artigo diz, não fornece informações sobre condições ou a vida na Terra primitiva. Embora em uma síntese abiótica ou não-enzimatica o ferro pode atuar como catalisador.
Por exemplo, um das ideias concorrentes com a de Miller vem do alemão Wächtershäuser que usa uma fonte interna de energia, a base de sulfetos de ferro (pirita de ferro) e outros minerais fazendo reações redox (redução e oxidação) de sulfuretos de metal para a síntese de moléculas orgânicas. Certamente teria ocorrido em uma fonte hidrotermal. As experiências com tais sulfuretos num meio aquoso a 100°C produziram um rendimento de dipéptidos (0,4% a 12,4%) e um menor rendimento de tripéptidos (0,10%)
– Huber, Claudia; Wächtershäuser, Günter (31 July 1998). “Peptides by Activation of Amino Acids with CO on (Ni,Fe)S Surfaces: Implications for the Origin of Life”. Science(Washington, D.C.: American Association for the Advancement of Science) 281 (5377): 670–672.
Essas áreas ai do BGB foram formadas em regiões oceânicas antigas (02) mas tem regiões vulcânicas metamorfoseadas, ou seja, re-cristalizadas e se forem datadas vão dar erro de data. O certo é datar a rocha vulcânica e não a metamorfizada. Ao que parece, é uma das poucas partes do Arqueano mais bem preservadas da crosta e pode fornecer dados sobre da Terra a 3 bilhões de anos, mas claro, a atividade tectônica cria todo este debate.
Tem muitas rochas vulcânicas lá. Tem as rochas ultramáficas, intrusivas rasas que são vulcânicas, tem as sedimentares, tem minerais de feldspatos alcalinos e regiões datadas em 3.2 bilhoes de anos representadas pelas rochas máfica e ultramáficas vulcânicas.
Lowe, R. David; Gary R. Byerly (2007). “An overview of the geology of the Barberton greenstone belt and vicinity:Implications for early crustal development”. Developments in Precambrian Geology 15.
Obviamente que há datas mais recentes, de 3.2 milhões de anos. Na verdade, não entendo como uma pessoa vem dizer que as datações estão erradas e citaram este artigo que você mandou. Ele ta falando do Pleistoceno que por si só já mata a ideia de Terra jovem criacionista.
De fato, isto pouco importa, um estudo feito em 2011 (ou seja, posterior a este que voce me mandou, que é de 2006) já deixou claro que regiões do BGB foram formadas pelo derretimentos parciais de rochas graníticas nas margens do cinturão e depois foram dobrados em um evento de dois estágios datado entre 3.26 e 3.22 Ga.
Van Kranendonk, Martin J. (2011). “Cool greenstone drips and the role of partial convective overturn in Barberton greenstone belt evolution”. Journal of African Earth Science 60: 346–352.
Portanto, há um conjunto geológico de evidencias diretas sobre a e evolução da Terra um pouco antes de 3 bilhões de anos, período na qual a vida já existia. Mas, as evidencias geológicas dão evidencias claras de crosta terrestre inicial, da química dos oceanos, da biota e atmosfera.
Lowe, R. David; Gary R. Byerly (2007). “An overview of the geology of the Barberton greenstone belt and vicinity:Implications for early crustal development”. Developments in Precambrian Geology 15
Obrigado pela resposta Rossetti, é que como eu disse, não entendo muito de geologia, mas eu sabia que como sempre criacionistas estavam sendo criacionistas, ou seja, pegando artigos cientificos fora de contexto e usando a seu favor.
Os caras são tão desonestos que ao inves de procurar evidencias para a tal terra
jovem, ficam lendo artigos e apontando como evidencias contra a evolução, e usam termos como “cronologia evolucionista” e outras falacias criacionistas.
O pior foi o cara usando o darwinismo.wordpress como referencia.
Otimo artigo e comentario Rossetti.
Isto se chama subversão. Quando voce torce, retorce, inverte e espreme uma afirmação para que ela diga o que ela nao diz e confirmar uma premissa pessoal….temos o costume de chamar isto de argumento do espantalho as vezes!!!
Subversão é o que a maioria dos posicionamentos opressores fazem. Eles não produzem absolutamente nada e se fundam em subverter o que é produzido pelos outros. Na política isto ocorre muito, especialmente entre os conservadores